Neue Details zum molekularen Postversand in der Zelle
Archivmeldung vom 04.11.2006
Bitte beachten Sie, dass die Meldung den Stand der Dinge zum Zeitpunkt ihrer Veröffentlichung am 04.11.2006 wiedergibt. Eventuelle in der Zwischenzeit veränderte Sachverhalte bleiben daher unberücksichtigt.
Freigeschaltet durch Thorsten Schmitt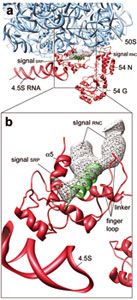
Wissenschaftlern des Berliner Max-Planck-Instituts für molekulare Genetik, der LMU München und der Universität Heidelberg ist es gelungen, mit bislang unerreicht hoher Auflösung neue Details des komplexen biologischen Prozesses der Proteinsortierung in der Zelle darzustellen.
Mit Hilfe von Kryo-Elektronenmikroskopie und Einzelpartikelanalyse konnten die Forscher erstmals im Detail sichtbar machen, wie Proteinketten beim Verlassen des Ribosoms von einem Signalerkennungsprotein erkannt werden. Dieser Vorgang läuft nach den Erkenntnissen der Wissenschaftler bei Bakterien und höheren Organismen nach dem gleichen Mechanismus ab [Nature, 29. Oktober 2006, Advance Online Publication]. Dieser Vorgang läuft nach den Erkenntnissen der Wissenschaftler bei Bakterien und höheren Organismen nach dem gleichen Mechanismus ab [Nature, 29. Oktober 2006, Advance Online Publication].
Die Sortierung bestimmter Proteine in der Zelle und der Transport
zu ihrem jeweiligen Bestimmungsort sind ein zentraler Schritt für die Funktion
aller Organismen. Die Mehrzahl der Proteine wird bereits während ihrer
Biosynthese ihrem späteren Einsatzort zugeordnet (kotranslationale
Translokation). Dies geschieht mit Hilfe eines molekularen Komplexes, der aus
einem Ribosom, also der Protein-Synthese-Maschine der Zelle, und einem
Signalerkennungsprotein (engl. signal recognition particle, SRP) besteht.
Das Schlüsselelement für die Proteinsortierung ist jedoch eine
Signalsequenz, welche sich am Anfang - Wissenschaftler sprechen vom N-terminalen
Ende - des vom Ribosom gebildeten Proteins befindet und quasi als "Postleitzahl"
in der Zelle fungiert. Das Signalerkennungsprotein (SRP) liest diese Sequenz,
sobald sie am Anfang einer gerade neu gebildeten Proteinkette das Ribosom
verlässt. Im nächsten Schritt bindet SRP an das Ribosom und leitet es unter
Beteiligung weiterer Komponenten an das endoplasmatischen Reticulums (ER)
weiter, wo die nächsten Schritte der Sortierung erfolgen.
Das
Wissenschaftlerteam der LMU München, der Universität Heidelberg und des Berliner
Max-Planck-Instituts für molekulare Genetik, denen im Rahmen des Berliner
UltraStrukturNetzwerk (USN) ein modernes Kryo-Elektronenmikroskop zur Verfügung
steht, konnten nun in bislang unerreichter Qualität auf molekularer Ebene
darstellen, wie die Signalsequenz durch SRP erkannt wird. Die Signalsequenz, die
"Adresse" des Proteins, bindet an eine spezielle Bindungstasche des SRP, die
sogenannte M-Domäne. Diese Bindung führt zu Umlagerungen / Strukturveränderungen
im SRP selber, woduch die Überführung der Signalsequenz an den
Translokon-Komplex eingeleitet werden (siehe auch MPG-Presseinformation [1]).
Immerhin etwa 30% aller Proteine der höher entwickelten Lebewesen wie z.B. des Menschen werden mit diesem Mechanismus sortiert - vor allem sekretorische Proteine, beispielsweise Antikörper, und Membranproteine, die unter anderem als Empfängermoleküle für neuronale Botenstoffe oder andere Signalmoleküle dienen. Dieser Vorgang findet bei Bakterien und Säugetierzellen in vergleichbarer Weise statt. Seine Aufklärung ist ein wichtiger Baustein zum Verständnis, auf welche Weise sekretorische Proteine bzw. Membranproteinen nach ihrer Bildung in der Zelle weitergeleitet werden.
Quelle: Pressemitteilung Max-Planck-Gesellschaft zur Förderung der Wissenschaften e.V.





