Selbstlernende Netzwerke lassen Forscher mehr sehen
Archivmeldung vom 07.12.2018
Bitte beachten Sie, dass die Meldung den Stand der Dinge zum Zeitpunkt ihrer Veröffentlichung am 07.12.2018 wiedergibt. Eventuelle in der Zwischenzeit veränderte Sachverhalte bleiben daher unberücksichtigt.
Freigeschaltet durch Manuel Schmidt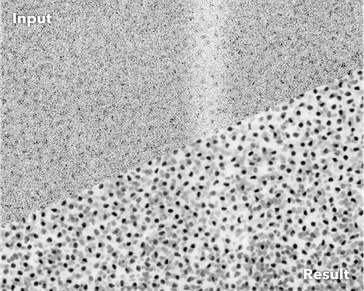
Moderne Mikroskope können mehrstündige 3D-Zeitrafferfilme von jeder einzelnen Zelle im sich entwickelnden Organismus aufnehmen. Genau wie bei normaler Fotografie benötigen Fluoreszenzmikroskope eine ausreichende Menge Licht, um dunkle und verrauschte Bilder zu vermeiden. Allerdings kann die für solche Filme benötigte Lichtmenge die oft untersuchten Modellorganismen wie Würmer, Fische und Mäuse schädigen.
Bisher ist die einzige Möglichkeit, diesen "ultimativen Sonnenbrand" zu vermeiden, kürzere Filme aufzunehmen oder aber die verwendete Lichtmenge zu reduzieren. Dadurch sind viele Biologen gezwungen, verrauschte und damit schwer interpretierbare Aufnahmen zu verwenden. Forscher um Florian Jug und Eugene W. Myers vom Zentrum für Systembiologie Dresden (CSBD) und vom Max-Planck-Institut für molekulare Zellbiologie und Genetik (MPI-CBG) haben nun eine inhaltssensitive Methode zur Bearbeitung von Mikroskopieaufnahmen entwickelt, die dieses Dilemma löst. Die selbstlernende Software namens CARE basiert auf künstlichen neuronalen Netzwerken und macht die in schlecht ausgeleuchteten Mikroskopiebildern verborgenen Inhalte sichtbar. CARE-Netzwerke sind in der Lage, hochaufgelöste Bilder herzustellen, auch wenn sie mit bis zu 60 mal weniger Laserlicht aufgenommen wurden. Damit ermöglicht CARE bildgebende Experimente, die bisher nicht denkbar waren. Die neue Methode ist frei verfügbar und so konzipiert, dass sie von jedermann genutzt und angepasst werden kann.
Fluoreszenz-Mikroskopie ist für die Grundlagenforschung im Bereich der Biomedizin unverzichtbar geworden. Die Technik kann die Position von fluoreszierenden Zellbausteinen in biologischen Geweben und Organismen sichtbar machen. In lebenden Proben können über viele Stunden hinweg dynamische Prozesse aufgezeichnet werden, so dass die Forscher zum Beispiel untersuchen können, wie Zellen während der Embryonalentwicklung Gewebe und Organe formen.
Die Qualität der Bilder hängt jedoch stark von der Menge des bei der Aufnahme verwendeten Lichts ab. Lichtverhältnisse, die zu qualitativ hochwertigen Bildern führen, verursachen unerwünschte Nebenwirkungen. Diese Nebenwirkungen sind als Phototoxizität bekannt und führen zu Veränderungen im Verhalten der Zelle; können für diese sogar tödlich sein. Darüber hinaus reagieren einige Organismen schon auf geringe Lichtmengen mit Muskelzucken, was die gewonnenen Daten ebenfalls unbrauchbar macht. Um diesen "ultimativen Sonnenbrand" der Phototoxizität zu vermeiden, müssen die Forscher die Gesamtmenge des verwendeten Lichts stark reduzieren. Das führt zu minderwertigen Bildern, die schwer zu analysieren sind.
Eine interdisziplinäre Forschergruppe am CSBD und MPI-CBG in Dresden hat nun ein Verfahren entwickelt, um hochwertige Bilder mit bis zu 60 mal weniger Licht zu erzeugen. Der neuartige Ansatz - CARE - ist eine selbstlernende, inhaltssensitive Bildrestaurierungs-Software, die auf künstlichen neuronalen Netzwerken basiert. Die Wissenschaftler kamen zu dem Schluss, dass man zwar keinen langen Film mit lauter hochwertigen Bildern aufnehmen kann ohne in die Phototoxizitäts-Falle zu laufen, es aber durchaus möglich ist, Schnappschüsse von Bilderpaaren aufzuzeichnen: je eins mit schlechter Lichtqualität und das andere mit ausreichend Licht, um sehr klare Bilder zu erzeugen. Diese Schnappschuss-Paare werden dann verwendet, um selbstlernenden CARE-Netzwerken beizubringen, die "versteckten" Inhalte in stark verrauschten Bildern sichtbar zu machen, also qualitativ hochwertige Bilder zu rekonstruieren. In ihrer kürzlich in Nature Methods veröffentlichten Studie zeigen die Forscher, dass CARE erfolgreich auf viele verschiedene Mikroskope, Experimente und Modellorganismen angewendet werden kann.
Martin Weigert, Erstautor und Mitglied der Myers Gruppe, sagt: "Ein wichtiger Nutzen unserer Methode ist, dass sie die Beobachtung von Zell- oder Gewebeprozessen unter sehr schwierigen Bedingungen ermöglicht, indem die Qualität der aufgenommenen Bilder deutlich verbessert wird“. Sein ehemaliger Kollege im Myers Team und Co-Autor der Studie, Loïc Royer, der mittlerweile seine eigene Gruppe am Chan Zuckerberg Biohub in San Francisco leitet, fügt hinzu: "Die Darstellung lebender Organismen erfordert oft Kompromisse. Mit CARE müssen Biologen solche drastischen Kompromisse nicht mehr eingehen. Unsere Methode macht bisher unmögliche bildgebende Experimente möglich."
"CARE ist ein Paradebeispiel für die Art von bahnbrechender Technologie, die ein interdisziplinärer Campus, wie unserer hier in Dresden-Johannstadt, hervorbringen kann. Informatiker, Physiker, Biologen und Chemiker des CSBD, des MPI-CBG und anderer DRESDEN-concept Institute haben eng zusammengearbeitet. Jeder hat sein spezielles Wissen eingebracht, um diesen wichtigen Fortschritt zu erzielen!", sagt Florian Jug, der an vielen Aspekten der Arbeit beteiligt war. Er schließt: "CARE öffnet Fenster, durch die wir die Prozesse, die biologisches Leben steuern, besser beobachten können. Wir sind gespannt, was kreative Köpfe auf der ganzen Welt nun mithilfe von CARE erforschen werden."
Quelle: Max-Planck-Institut für molekulare Zellbiologie und Genetik (idw)







